Pourquoi identifier les épitopes conformationnels ?
L’objectif de l’épitope mapping est d’identifier l’épitope contre lequel est dirigé un anticorps monoclonal.
Les épitopes conformationnels sont formés à partir de résidus qui sont distants dans la séquence primaire mais réunis par les repliements de la protéine d'intérêt. La détermination des épitopes conformationnels représentent un enjeu majeur pour les acteurs du monde de l'anticorps. Cependant, les techniques habituelles pour la détermination d'épitopes linéaires ne sont pas opérantes.
Agro-Bio a ainsi développé une méthode robuste d'identification des épitopes conformationnels d'un anticorps par des techniques de protéolyse in situ avec la formation d'un complexe antigène-anticorps immobilisé, suivi d'analyses par spectrométrie de masse.
La méthode d'Agro-Bio pour l'identification d'épitopes conformationnels

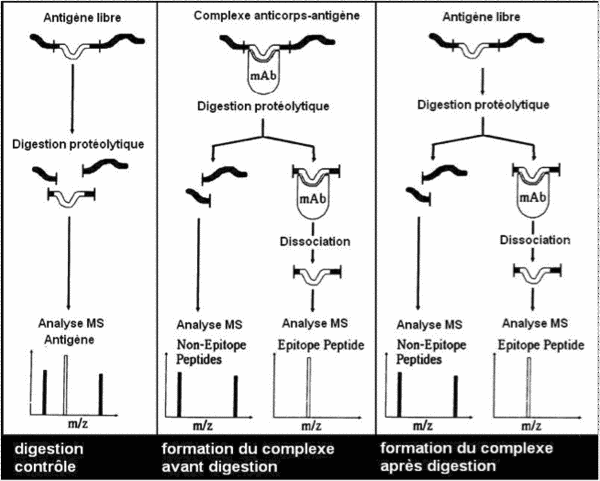
Les différentes étapes de l'épitope mapping
Notre méthode combine :
- une étape de digestion protéolytique
- une étape d’isolation des peptides de l’épitope par chromatographie d’affinité
- l’analyse finale des fragments de l’épitope par spectrométrie de masse.
Deux approches sont possibles :
- le complexe Antigène/Anticorps est formé lors de la chromatographie d’affinité et la digestion du complexe est réalisée au sein de la colonne d’affinité
- la digestion de l’antigène est effectuée avant la formation du complexe Antigène/Anticorps au sein de la colonne d’affinité.
La recherche de cette séquence d’intérêt, proposée par Agro-Bio, est basée sur la protection de l’épitope contre une digestion enzymatique par la formation du complexe Antigène/Anticorps.
Après passage sur chromatographie d’affinité et digestion, les fragments de l’épitope sont élués puis séquencés par spectrométrie de masse.
Les avantages de la cartographie épitopique par AGRO-BIO
Choix des enzymes, Purification, Spectrométrie de masse, trois expertises simultanées :
Une étude in silico est lancée pour simuler l’action des enzymes et reproduire la digestion de la protéine d’intérêt. Idéalement, l’enzyme de digestion doit couper au plus près des frontières de l’épitope. Le choix des enzymes de digestion doit donc être fait en fonction de la fréquence de coupure des enzymes dans la séquence de la protéine recombinant (au moins 2 enzymes protéases sont recommandées). En fonction de la disponibilité de la structure 3D de la protéine d’intérêt, il sera possible d’y confronter les résultats que nous obtiendrons et déterminer les peptides « candidats » les plus intéressants.
Une digestion enzymatique en solution de Antigène et Anticorps est ensuite réalisée, afin d’obtenir tous les peptides, y compris les séquences épitopiques, ainsi que les peptides non épitopiques de la même protéine. Le succès de la réaction de protéolyse est ensuite contrôlé en spectrométrique de masse pour avoir une cartographie des peptides.
Pour avoir la détermination de l'épitope, une colonne de chromatographie d'affinité est utilisée avec l’immobilisation de l’anticorps d’intérêt. La formation du complexe Anticorps-Antigène dans la colonne est suivie d'une digestion enzymatique (basée sur les informations de l'étude in silico). Tous les peptides non liés sont lavés et les peptides épitopes d'intérêt récupérés puis séquencés par spectrométrie de masse à haute résolution.

